Des questions et des sources multiples
S’intéresser au phénomène infectieux dans le passé recouvre une large diversité de problématiques, depuis l’évolution moléculaire et la diffusion de tel ou tel agent pathogène, jusqu’aux conséquences sanitaires sur les communautés anciennes l’ayant expérimenté, leur compréhension du phénomène et leurs éventuelles tentatives d’y faire face. Cela concerne aussi les conditions environnementales et les interactions ayant rendu ces phénomènes possibles, ce qui peut inclure les activités humaines.

Les sources qu’il est possible de mobiliser sont tout aussi diversifiées : traités médicaux anciens et chroniques relatant la survenue d’une épidémie, registres paroissiaux mentionnant les causes de décès, vestiges archéologiques comme la diffusion des léproseries au Moyen-Âge, biorestes macroscopiques fossiles ou subfossiles comme des ossements, biorestes microscopiques comme des formes de résistance parasitaire, ou moléculaires comme des coproantigènes ou des fragments d’ADN signant la présence d’un agent infectieux en particulier dans l’échantillon considéré.
A l’intersection des sciences humaines et naturelles
Bien entendu, plusieurs types de sources peuvent communément servir à investiguer un même phénomène infectieux, par exemple dans le cas de la deuxième pandémie de peste pour laquelle existent de nombreuses archives historiques. On peut en cela citer notamment les recensements effectués dans les localités du sud de la France et pré-datant l’épisode pesteux de 1720-1722, et les actes de sépultures post-datant celui-ci, comme à Aubagne ou Martigues, ainsi que les sépultures de masse mises en évidence dans les mêmes localités par l’archéologie comme celles dites « du Délos » et « des Capucins de Ferrières » à Martigues (Signoli, 2018, p. 73-74, 103). A l’aune de ces sources, on constate tout à la fois la temporalité des flambées épidémiques locales, l’identité des victimes et leur distribution dans la population en termes d’âge ou de sexe, ainsi que les méthodes employées pour la gestion des corps par les communautés touchées.

Pour les époques pré-datant l’existence d’archives modernes qui autorisent l’évaluation fiable de la ponction démographique occasionnée par la survenue d’une épidémie, l’existence de chroniques extensives ou autres témoignages écrits peut néanmoins permettre d’apercevoir la réponse de communautés anciennes à ces phénomènes, à plus fortes raisons de leurs autorités. Ce fut par exemple le cas lors de la première pandémie de peste, dite « de Justinien », au 6ème siècle de notre ère et dont la gestion, d’une part sous l’égide du pape Grégoire 1er à Rome, et d’autre part de l’empereur Justinien à Byzance, a fait l’objet de témoignages écrits (Bertrand, 2018). Là encore, il est permis d’aborder le phénomène au travers de différentes problématiques, mais également de différents proxies, puisque l’agent causal de la « peste » de Justinien a été identifié par la paléomicrobiologie comme étant effectivement celui de la peste, la bactérie Yersinia pestis (Harbeck et al., 2013). La chose n’est cependant pas toujours aussi claire, et nombre d’épidémies historiquement connues n’ont pas encore fait l’objet d’identification formelle, consensuelle ou non ambiguë de leur agent causal. On peut évoquer en cela la « peste » d’Athènes qui frappa la ville en 430 avant notre ère (du vivant de Périclès), ou encore la « peste » antonine, qui toucha l’empire romain dans la deuxième moitié du 2ème siècle de notre ère, à l’époque de la dynastie des Antonin, bien qu’il existe parfois quelque candidat crédible à cette identification (Harper, 2019, p.169). Et ceci est sans parler des épidémies, pandémies et circulations endémiques n’ayant jamais fait l’objet de témoignages écrits ou dont ceux-ci ne nous sont pas parvenus.
Focus sur l’approche paléogénomique
Dans bien des cas, seule l’identification moléculaire d’un agent infectieux permet d’attester de sa présence non spéculative en un lieu donné dans le passé lointain, à l’exception de ceux produisant également des biorestes par exemple identifiables sous microscopes, comme certains parasites gastro-intestinaux (Le Bailly et al., 2021). Plus précisément, l’obtention de séquences d’ADN ancien suffisamment longues et spécifiques permet non seulement cette identification formelle, mais également l’étude de la diversité et de l’évolution de ces organismes.
Cela concerne par exemple l’agent de la peste, dont nous avons déjà parlé (ici), mais également d’autres agents bactériens pour lesquels nous commençons à posséder des génomes anciens complets. On peut ainsi citer l’agent de la tuberculose (TB), Mycobacterium tuberculosis, qui est l’une des infections les plus prévalentes au sein de l’espèce humaine et l’une des plus étudiées en paléomicrobiologie à ce jour ainsi que la première à avoir fait l’objet de recherches paléogénétiques (Spigelman et Lemma, 1993). Les données paléogénétiques justement acquises dans le courant des années 90 ont rapidement montré l’ancienneté de la circulation de cette infection au sein des communautés humaines dès avant -3000 ans (eg. Crubézy et al., 1998). Par la suite, le développement des technologies de séquençage a notamment permis d’éclaircir la question de la migration transocéanique de la TB et de son arrivée sur le continent américain. Effectivement, l’étude de génomes anciens complets provenant d’Amérique du Sud obtenus depuis des restes humains archéologiques présentant des signes osseux de tuberculose a permis non seulement d’en détecter l’agent causal, mais en plus de montrer sa plus grande proximité phylogénétique avec les souches infectant communément les pinnipèdes que celles infectant les humains. Il était donc permis de voir là une origine zoonotique de la TB aux Amériques et sa dissémination aux communautés humaines pré-Contactes dès avant l’introduction de nouvelles souches lors de l’arrivée des européens (Bos et al., 2014).
Comme nous l’avions déjà évoqué pour l’agent de la peste, ce type de travaux nous renseigne non seulement sur la diffusion de ces maladies mais également sur leur évolution, notamment celle de leur virulence. A ce propos, l’obtention de données paléogénomiques concernant la bactérie responsable de la lèpre, Mycobacterium leprae, a permis de montrer la quasi absence de différences génétiques entre les souches modernes et anciennes, remettant donc en cause l’hypothèse selon laquelle la disparition de cette maladie en Europe avait à voir avec les mutations génétiques de son agent causal, mais plutôt avec des raison exogènes, comme les conditions de vie ou l’immunité changeantes des hôtes humains infectés (Schuenemann et al., 2013).
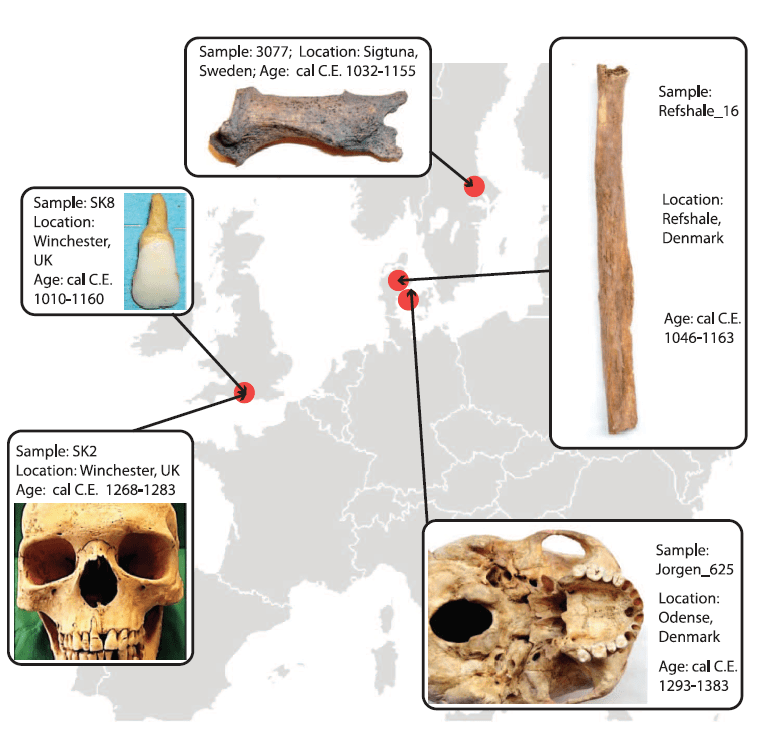
Le rôle des migrations transocéaniques tout autant que des transmissions entre hôtes animaux et humains est bien illustré par les travaux récents concernant les salmonelles responsables de fièvres typhoïdes et paratyphoïdes, respectivement Salmonella enterica Typhi et S. enterica Paratyphi. La comparaison d’une souche identifiée dans les restes d’un individu décédé au 13ème siècle en Norvège avec les souches touchant les animaux domestiques actuels a effectivement permis de suggérer que la souche paratyphoïde aujourd’hui spécifique des humains avait probablement émergé en Europe à la faveur de la domestication des suidés dans la région il y a environ 4000 ans (Zhou et al., 2018). Plus récemment encore, la comparaison de plusieurs génomes anciens de S. enterica a permis de mettre en évidence le rôle du processus de néolithisation dans l’adaptation de souches initialement non spécifiques à l’hôte humain (Key et al., 2020), ainsi que celui des routes d’échanges comme voies de diffusion, par exemple la propo-route de la soie durant l’Âge du Bronze (Wu et al., 2021). De l’autre côté de l’Atlantique au Mexique, c’est la souche responsable de la fièvre typhoïde qui a pu être détectée au 16ème siècle, au moment où se produisait une épidémie de cocoliztli documentée historiquement, quelques années après le premier contact avec les européens dans la région (Vågene et al., 2018).
De tels travaux ne concernent cependant pas uniquement des agents bactériens, et on peut citer les exemples d’eucaryotes comme l’agent du Paludisme, le Plasmodium, un protozoaire sanguin, qui a pu être détecté chez des individus décédés en Italie aux 1er et 2ème siècles de notre ère à l’époque romaine (Marciniak et al., 2016), ainsi que dans des échantillons médicaux de la première moitié du 20ème siècle. En effet, la recherche d’ADN ancien d’agents pathogènes peut être réalisée sur une grande diversité d’échantillons, de part leur nature, provenance ou ancienneté, et non seulement des ossements. Ici, les chercheurs ayant mis en évidence le génome mitochondrial de la souche européenne aujourd’hui éteinte du paludisme (la maladie a été éradiquée d’Europe au milieu du 20ème siècle), ont noté sa grande proximité avec la souche Américaine, et ont donc proposé que celle-ci avait été introduite dans le Nouveaux Monde post-Contact (Gelabert et al., 2016).
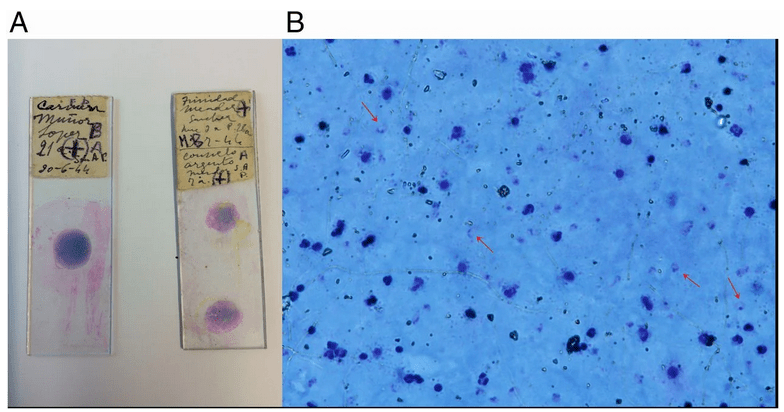
Enfin, ce type de recherche s’intéresse également aux particules virales, et on peut citer le cas du virus de la variole (le VARV), qui a pu être séquencé depuis les restes d’individus décédés 20ème et au 17ème siècles, montrant au passage l’évolution extrêmement récente de ce virus (Duggan et al., 2016).
In fine
Ce petit tour d’horizon non exhaustif permet d’appréhender l’univers que constitue à elle seule l’investigation des phénomènes infectieux dans le passé. Les problématiques abordables sont extrêmement nombreuses, impliquant tout à la fois les sciences de l’Homme et des sociétés, les sciences de la vie et de l’environnement, et tout autant de sources et de méthodes, depuis la traduction de chroniques en langues anciennes, au dépouillement d’actes de décès, et en passant par la recherche de fragments d’ADN et l’examen de restes ostéologiques ou momifiés. En dépit de l’accent mis ici sur l’approche paléogénomique, appréhender les dynamiques infectieuses du passé à l’actuel est donc une entreprise hautement collaborative et pluridisciplinaire, à laquelle toute une diversité de spécialistes peut participer.
Références :
- Bertrand, F. (2018). Les autorités civiles et religieuses face à la première pandémie pesteuse, dite « Peste de Justinien » (541-767) in Séguy, I., Ginnaio, M., Buchet, L., (ed) Les conditions sanitaires des populations du passé. Environnement, maladies, prophylaxies et politiques publiques. Edition APDCA – Antibes, p. 133-150
- Bos, K. et al., (2014). Pre-Columbian mycobacterial genomes reveal seals as a source of New World human tuberculosis. Nature, 514(7523), 494–497. doi: 10.1038/nature13591
- Crubézy, E. et al., (1998). Identification of Mycobacterium DNA in an Egyptian Pott’s disease of 5400 years old. , 321(11), 0–951. doi:10.1016/s0764-4469(99)80009-2
- Duggan, A. T. et al., (2016). 17th Century Variola Virus Reveals the Recent History of Smallpox. Current biology : CB, 26(24), 3407–3412. doi: 10.1016/j.cub.2016.10.061
- Gelabert, P. et al., (2016). Mitochondrial DNA from the eradicated European Plasmodium vivax and P. falciparum from 70-year-old slides from the Ebro Delta in Spain. Proceedings of the National Academy of Sciences of the United States of America, 113(41), 11495–11500. doi: 10.1073/pnas.1611017113
- Harper, K. (2019). Comment l’Empire Romain s’est effondré. Les climat, les maladies et la chute de Rome. La Découverte, Paris. 544 p.
- Harbeck, M. et al., (2013). Yersinia pestis DNA from skeletal remains from the 6th century AD reveals insights into Justinianic Plague. PLoS pathogens, 9(5), e1003349. doi: 10.1371/journal.ppat.1003349
- Key, F. M. et al., (2020). Emergence of human-adapted Salmonella enterica is linked to the Neolithization process. Nature ecology & evolution, 4(3), 324–333. doi: 10.1038/s41559-020-1106-9
- Le Bailly, M. et al., (2021). Accessing Ancient Population Lifeways through the Study of Gastrointestinal Parasites: Paleoparasitology. Applied Sciences, 11(11), 4868. MDPI AG. doi: 10.3390/app11114868
- Marciniak, S. et al., (2016). Plasmodium falciparum malaria in 1st-2nd century CE southern Italy. Current biology : CB, 26(23), R1220–R1222. doi: 10.1016/j.cub.2016.10.016
- Schuenemann, V. J. et al., (2013). Genome-wide comparison of medieval and modern Mycobacterium leprae. Science (New York, N.Y.), 341(6142). doi: 10.1126/science.1238286
- Signoli, M., (2018). La Peste Noire. Que sais-je / Humensis, Paris, 128 p.
- Spigelman, M., & Lemma, E. (1993). The use of the polymerase chain reaction (PCR) to detect Mycobacterium tuberculosis in ancient skeletons. International Journal of Osteoarchaeology, 3(2), 137–143. doi:10.1002/oa.1390030211
- Tzortzis, S., & Signoli, M. (2009). Les tranchées des Capucins de Ferrières (Martigues, Bouches-du-Rhône, France). Un charnier de l’épidémie de peste de 1720 à 1722 en Provence. Comptes Rendus Palevol, 8(8), 749–760. doi:10.1016/j.crpv.2009.06.005
- Vågene, Å. J. et al., (2018). Salmonella enterica genomes from victims of a major sixteenth-century epidemic in Mexico. Nature ecology & evolution. doi: 10.1038/s41559-017-0446-6
- Wu, X., et al., (2021). A 3,000-year-old, basal S. enterica lineage from Bronze Age Xinjiang suggests spread along the Proto-Silk Road. PLoS pathogens, 17(9), e1009886. doi: 10.1371/journal.ppat.1009886
- Zhou, Z. et al., (2018). Pan-genome Analysis of Ancient and Modern Salmonella enterica Demonstrates Genomic Stability of the Invasive Para C Lineage for Millennia. Current biology : CB, 28(15), 2420–2428.e10. doi: 10.1016/j.cub.2018.05.058
